前言
The Medical Imaging Interaction Toolkit (MITK)是一个免费的开源软件,用于开发交互式医学影像处理软件。最近突然安排我做相关的一些工作,首先就要从编译开始,当然官网也有编译好的版本,可以直接下载使用。本来在Windows上编译这种开源的软件就很麻烦,在加上github上的东西下载巨慢,常常出错,折腾了好久才编译完成,这里就记录一下踩过的那些坑。
准备工作
- Visual Studio 2017
- CMake (>=3.19)
- Qt 5.12.10 (>=5.12.9)
- Python3
- Git
- OpenSSL 安装包
- Doxygen
- MITK
- MITK-Diffusion
我这里使用的是Visual Studio 2017版本,2019应该也可以。
Qt需要安装5.12.9以上的版本,官方编译似乎用的5.12.10,所以我这里也选择5.12.10版本,安装过程中尽量把所有组件都选上,因为编译时会使用到很多组件。
CMake,Python,OpenSSL都选择64位版本安装。
最后就是用git克隆下MITK和MITK-Diffusion的仓库。
由于MITK在编译的过程中会下载一些第三方的软件包,所以要能克隆github上的仓库才行,最好有梯子,我就是这里卡了很久。
CMake 相关配置
- 在MITK仓库目录下新建build文件夹(名字可以随意),然后打开cmake GUI工具,填写
source code和build文件夹路径 - 点击Configure按钮,第一次会弹出对话框选择编译器,根据需要配置即可,这里选择msvc-2017,x64
- 此时会出现错误提示,找不到Qt,需要手动设置一下Qt5的路径,找到
Qt5_DIR项,在Value中填写路径,例:D:/Qt/Qt5.12.10/msvc2017_64/lib/cmake/Qt5,再次点击Configure按钮 - 检查是否所有的安装路径都正确被检测到(Qt,OpenSSL),确保没有选项是红色
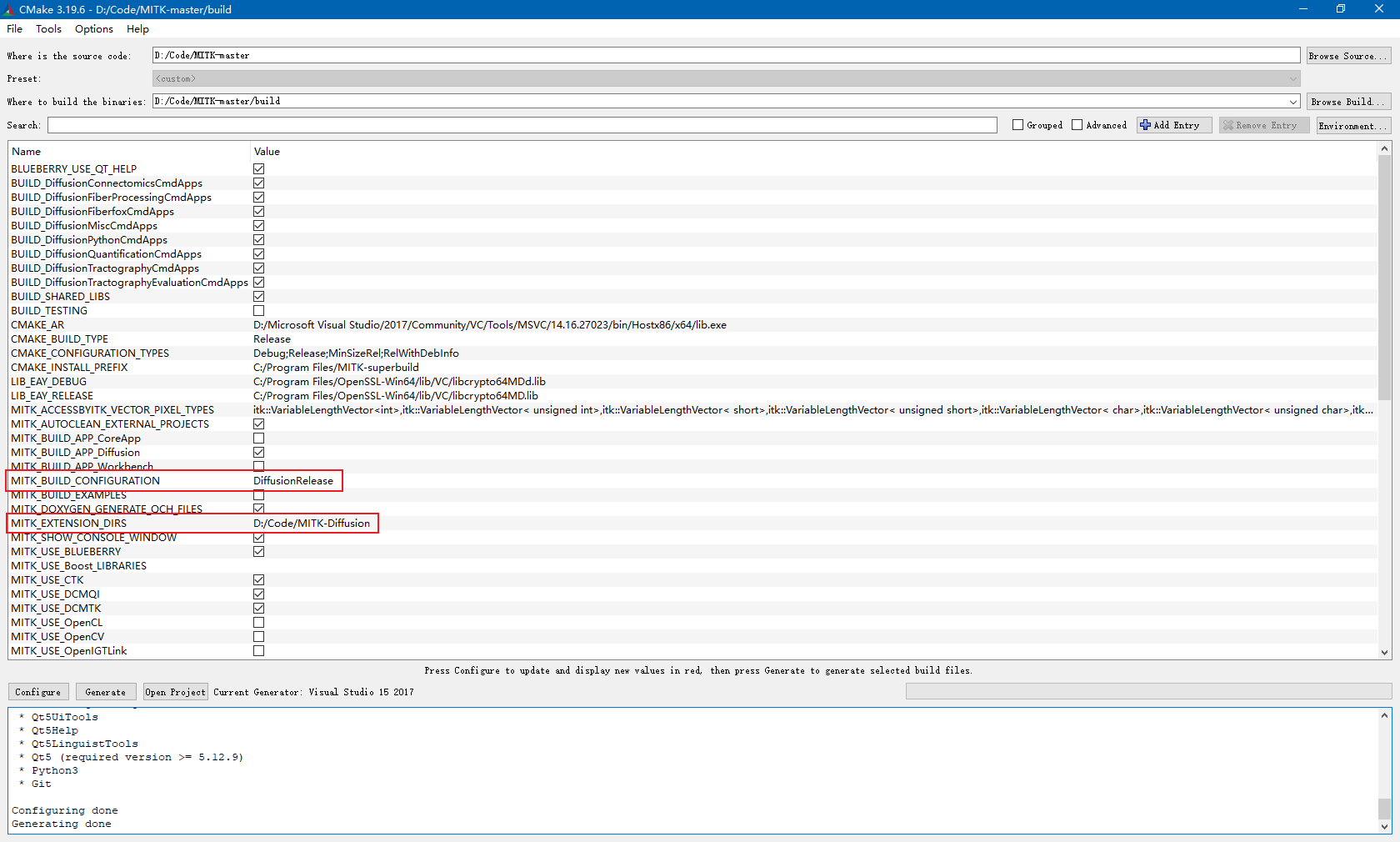
- 如果只需要编译MITK,那么就可以直接跳到第9步
- 找到
MITK_EXTENSION_DIRS选项,在Value中填写MITK-Diffusion仓库的路径,再次点击Configure按钮 - 找到
MITK_BUILD_CONFIGURATION选项,Value设置为DiffusionRelease,再次点击Configure按钮 - 此时下方输出会报错,提示找不到
NumPy,需要先安装python库NumPy,打开终端,执行pip3 install --user NumPy,完成后再次点击Configure按钮,确保没有错误,没有选项是红色 - 点击Generate按钮,下方输出
Generating done之后,点击Open Project按钮
附上我的配置:
编译
直接编译ALL_BUILD项目即可,但此时编译可能会有一堆莫名奇妙的错误,可以先到MITK仓库目录下找到CMakeExternals目录,然后将里面所有的文件换行符改为Windows下的换行符(CRLF),可以使用VS Code或者Notepad++等工具,过程会有点枯燥。
确保网络可用,最好有国外朋友帮忙,因为编译过程中会下载很多github上的仓库,没有国外朋友帮忙很容易出错。编译ALL_BUILD项目,第一次编译会非常慢,通常需要几个小时,建议先去忙点其它事情。
编译过程中经常会遇到警告被视为错误,没有生成object,导致编译出错,双击错误,打开错误文件,然后再找到错误文件的位置,用记事本打开,选择文件→另存为,选择保存编码为
Unicode,再次编译。在编译
MITK-Diffusion的过程中,可能会遇到一些类型转换的报错,如无法将itk::Point转换为mitk::PointSet::PointType等,可能一些编译器能通过,但msvc会报错,只需要稍微修改一下,将出错的参数强制转换成对应类型即可,如:mitk::PointSet::PointType(itkPoint)。
重复3、4若干次,直到编译成功。
编译后的可执行文件在build/MITK-build/bin目录下。